1 介绍
1.1 参考文章
在生物信息学的助力下,科研人员不断探索着癌症转录组学的奥秘,以期发现能够早期诊断、指导治疗和预测疾病进程的生物标志物。本教程参考已发表的文章(Yang 等 2023)的研究方法,该文章深入探讨了慢性阻塞性肺病(COPD)晚期的潜在诊断基因生物标志物,为科研人员提供了宝贵的经验和启示。
1.2 HCC项目流程
该文章已发表了,请勿使用该初稿投递任何期刊,它仅作为学习的参考材料。
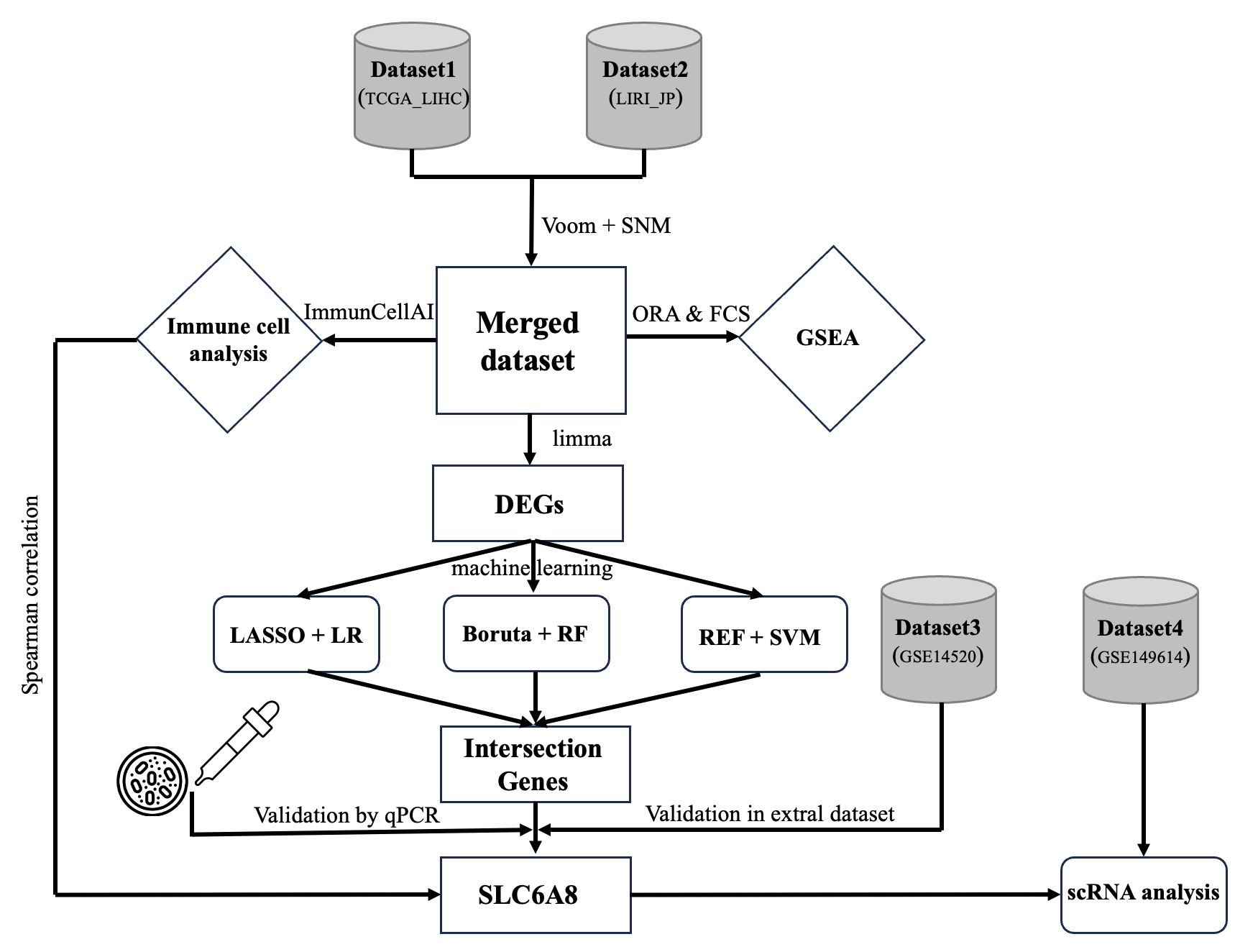
本书围绕癌症生信实战工作流分为以下几个部分:
- 基础知识。
- 数据准备。
- 差异分析。
- 功能分析。
- 浸润分析。
- 标记筛选。
- 关联分析。
- 单细胞分析。
- 撰写文章。
1.3 数据地址
为了方便用户进行本教程中的生物信息学分析,已将所需的所有输入数据和预期的输出数据整理并上传至百度网盘。用户可以通过访问指定的百度网盘链接,下载所需的数据集,以便在本地环境中进行后续的分析和验证。请确保在下载和使用数据时,遵循相关的版权和引用规定。
购买了该教程的用户,自动获得本教程所需要的所有数据。
1.4 基础知识
该部分旨在为初学者提供生物信息学分析所需的基本工具、语言、数学及数据库知识的概览。以下是各部分的简要介绍:
- 软件工具
本教程推荐使用R和RStudio作为主要的生物信息学分析工具。
- R语言基础
在进行生物信息学分析之前,掌握R语言的基础知识是至关重要的。
- 数学基础
数学是生物信息学分析的核心。
- 数据库基础
生物信息学分析离不开数据的支持。
1.5 数据准备
该部分详细阐述了一系列数据处理流程,旨在确保数据质量,为后续的深入分析奠定坚实基础。以下是对各个步骤的详细书面化处理:
数据收集
数据预处理
数据对象转换
数据校正
数据汇总
1.6 差异分析
差异分析limma
火山图
热图
1.7 功能分析
GO term富集分析
KEGG pathway富集分析
GSVA富集分析
1.8 浸润分析
免疫浸润分析
免疫细胞数据分析
1.9 标记筛选
在该部分中,我们采用了三种不同的特征筛选与机器学习算法模式来识别关键的标记基因。
LASSO+LR
Boruta+RF
REF+SVM
交集特征
验证特征
1.10 关联分析
通过关联分析核心特征与免疫浸润细胞,我们可以进一步理解这些特征在肿瘤微环境中的功能和作用,以及它们如何影响免疫浸润细胞的数量和功能。
1.11 单细胞分析
在该部分中,我们对原始单细胞转录组数据进行了详尽的处理和分析。
单细胞数据处理
单细胞数据标准
单细胞聚类分析
单细胞细胞识别
核心特征单细胞表达